物种 Beta 多样性
物种 Beta 多样性
距离和相似性
统计学上任何满足非负、自反和三角不等式的计算结果都可以称作距离,距离用来描述两个统计学对象的远近程度 [1],对象可以是坐标轴上的一个点或者多个点组成的群落。距离越远表明对象之间相差越大。
当两个对象相近的属性越多表明两个对象越相似。如正多边形的边数和边长两个属性,若两个正多边形边数相等,则两者相似,若边数和边长都相等则两者全等。对于两个群落,群落的物种组成就是他们的属性,两个群落共有的物种数量越多,共有物种的相对丰度约接近,两个群落的相似性越大。相似性通过相似性指数描述,相似性指数越高则样本越相似。
请注意
所有的相似性指数都可以转换成对应的距离,但并非所有的距离都可以转换成相似性指数。
Beta 多样性的本质
Beta 多样性 [2](Beta diversity)利用各样本丰度信息(基因、物种或功能)来计算样本间距离或相似性,通过距离反映样本(组)间(between-sample)或生境间(between-habitat diversity) 是否具有显著的微生物群落差异,又称生境间的多样性。
简单来说,Beta 多样性分析就是看样本之间的差异。
双零问题
微生物数据中经常出现某个微生物在两个样品中都没有检测到的情况,此时物种缺失(双零数据)的含义决定了它能否作为两个样本相似性的判断依据。有两种情况:
- 两个采样地点都不符合该物种的生存条件,或者该物种从未扩散到此地。
- 采样时样品中恰好没有包含该物种,或者该物种在样品中的含量过低丰度过低无法被注释出来。
双零数据在微生物生态中常常出现,如果双零数据的含义相同,则双零数据可以作为判断两者相似的依据。但宏基因组项目本身就是在探索未知的环境,因此双零数据在不同样品中的含义通常无法确定是否相同,并且随着被检测物种数量的增加,双零数据在样品间出现的概率也逐渐增加,这种不确定性也在增加。
对称和非对称被用来描述上述的双零问题,如双零数据含义相同则可以作为相似性判断的依据,称为对称,否则为非对称。在大部分情况下应该优先选择非对称,除非可以确定零数据的含义相同。
Beta 多样性度量
宏基因组项目通常采用欧式距离 [3](Euclidean distance)、Bray-Curtis 相异性指数(Bray-Curtis 距离)[4](Bray-curtis distance)和 JSD 距离(Jensen-Shannon divergence)来衡量样本(组) 间差异。多个样本之间互相计算距离,将距离信息放置在表格中组成一个距离矩阵,是 Beta 多样性分析的基础。
其中
欧式距离是对称指数(双零数据含义相同),另外两个为非对称指数。因此当样品对中有很多零值时应该谨慎使用欧式距离。
欧式距离
欧几里得距离是多变量分析中经常使用的一种距离,其计算公式如下:
、:两个样本
:样本中第 n 个物种
: 样本中的第 个物种的丰度
: 样本中的第 个物种的丰度
根据公式可以得出,欧氏距离的大小取决于输入的丰度大小,其的取值范围为 。欧式距离越大表明两个样本差异越大。
Bray-Curtis 距离
Bray-Curtis 距离是计算微生物丰度差异最常用的距离之一,但是 Bray-Curtis 的计算方法并不符合距离定义中的三角不等式规定,因此它并不是严格意义上的距离,其准确的叫法是 Bray-Curtis 相异性指数,但为了符合现有的称呼,本报告也将其称作 Bray-Curtis 距离。
在计算 Bray-Curtis 距离时,在两个样本中都没有检测出来的样本会被忽略掉。其计算公式如下:
、:两个样本
:比较两个样本中每个物种的丰度,取所有物种相对较低的丰度求和。
: 样本中所有物种的丰度之和
: 样本中所有物种的丰度之和
当使用相对丰度数据时,公式可以简化成
根据公式可以得出,Bray-Curtis 距离的取值范围为 ,当 时表明两个样本的物种组成完全一致,当 时表明两个样本中不存在共有物种。BC 值越小,表明两个样本的相似性越高,差异越小。
JSD 距离
JSD 距离(Jensen–Shannon divergence,JSD 散度)是从 KL 散度(Kullback–Leibler divergence)演变过来的用来描述概率分布的两样本相似差异的距离指标。
JSD 距离也是非对称指数,因此在两个样本中都没有检测到的样本会被忽略掉。其计算公式如下
其中, 的计算规则
的计算规则
:第一个样本的丰度矩阵
:第一个样本中第 个物种的丰度
:第二个样本的丰度矩阵
:第二个样本中第 个物种的丰度
根据公式可以得出,JSD 距离的取值范围为 ,JSD 值越小,表明两个样本的相似性越高,差异越小。
Beta 多样性描述的是样品之间的差异,使用样本的丰度数据或者相似性指数和距离进行描述。在具体实验中分组样本之间的差异如何,差异是否显著,关系到 实验分组 / 施加处理 是否有效,如与比较组滤材相比,使用某种新型过滤材料是否能显著改善附着细菌群落结构,从而提高滤池中氮磷的代谢。
然而,无论是丰度数据还是相似性和距离,物种组成数据均属于多变量(多维)样本。要对高维数据进行分类和可视化时,需要用到降维方法将高维的数据映射到 2 维平面或者 3 维立体空间内,这样我们才能直观地看出不同样本之间的距离和相似性。按照使用数据和统计方法的不同,常用的降维方法有 PCA 分析、PLS-DA 分析、PCoA 分析、NMDS 分析,它们的比较如下表:
| PCA | PLS-DA | PCoA | NMDS | |
|---|---|---|---|---|
| 中文名 | 主成分分析 | 最小二乘法判别分析 | 主坐标分析 | 非度量多维尺度法 |
| 是否监督 | 无监督 | 有监督 | - | - |
| 输入数据 | 丰度矩阵 | 丰度矩阵 | 距离矩阵 | 距离矩阵 |
| 坐标轴权重 | 有 | 有 | 有 | 无 |
注:
是否监督:有监督,考虑分组;无监督,不考虑分组
坐标轴权重:坐标轴对变量的解释度是否相同,解释度不相同的有坐标轴权重,否则没有坐标轴权重
PCA 分析
PCA [5](Principal Components Analysis) 主成分分析,也称主分量分析或主成分回归分析法。PCA 降维的思想是首先减少数据集的维数,同时还保持数据集对方差贡献最大的特征,最终使数据直观呈现在二维坐标系。
PCA 的主要步骤如下:
(1)对数据进行归一化处理;
(2)计算归一化后的数据集的协方差矩阵;
(3)计算协方差矩阵的特征值和特征向量;
(4)保留最重要的 k 个特征(通常 k 要小于 n);
(5)找出 k 个特征值相应的特征向量;
(6)将 m * n 的数据集乘以 k 个 n 维的特征向量的特征向量(n * k),得到最后降维的数据。
PLS-DA 分析
PLS-DA(Partial Least Squares Discriminant Analysis),即偏最小二乘法判别分析,是多变量数据分析技术中的判别分析法,经常用来处理分类和判别问题。通过对主成分适当的旋转,PLS-DA 可以有效的对组间观察值进行区分,并且能够找到导致组间区别的影响变量。
PLS-DA 采用了经典的偏最小二乘回归模型,其响应变量是一组反应统计单元间类别关系的分类信息,是一种有监督的判别分析方法。因无监督的分析方法(PCA) 对所有样本不加以区分,即每个样本对模型有着同样的贡献,因此,当样本的组间差异较大,而组内差异较小时,无监督分析方法可以明显区分组间差异;而当样本的组间差异不明晰,而组内差异较大时,无监督分析方法难以发现和区分组间差异。另外,如果组间的差异较小,各组的样本量相差较大,样本量大的那组将会主导模型。有监督的分析(PLS-DA)能够很好的解决无监督分析中遇到的这些问题。
PCoA 分析
多维尺度分析 [6](Multidimensional scaling,MDS) 是基于样本间相似性距离矩阵降维来可视化样本位置的分析方法,PCoA 也称为经典多维尺度分析。PCoA 分析常用的输入矩阵有欧式距离矩阵,Bray-Curtis 相异性矩阵,JSD 散度矩阵等。
NMDS 分析
NMDS [7](Non-metric Multidimensional Scaling)非度量多维尺度法是一种将多维空间的研究对象(样本或变量)简化到低维空间进行定位、分析和归类,同时又保留对象间原始关系的数据分析方法。适用于无法获得研究对象间精确的相似性或相异性数据,仅能得到他们之间等级关系数据的情形。检验 NMDS 结果的优劣用胁强系数(Stress 值)来界定,它表示了 2 维空间中点的距离与多维空间中点的距离的差异,其计算公式如下
通常认为当 Stress < 0.2 时,可用 NMDS 的二维点图表示,其图形有一定的解释意义;当 Stress < 0.1 时,可以认为是一个好的排序;当 Stress< 0.05 时,具有很好的代表性。
统计检验
多维数据的统计检验
微生物群落丰度数据是多维数据,其中一个物种表示一个数据维度。样本之间距离可以通过降维,在一个平面上表示,但他们是否真的存在差异,需要根据统计检验结果判断。常见的情况是:经过降维样品在二维平面上标注出来,但不同分组的样本之间存在部分重叠,此时在图中是不能判断分组间是否存在差异,需要有统计检验结果才能判断。
对一个因变量(一维)数据的统计检验称为(一元)方差分析,它是基于数据本身。类似地,对高维度数据样本的统计检验称为多元方差分析,它是以距离矩阵为基础,也就是将多个变量根据不同的距离算法先计算距离矩阵,然后对距离矩阵进行统计检验。
进行统计检验有助于回答:
- 自变量 (因素)的变化是否对因变量 (试验指标)有显着影响?
- 因变量之间有什么关系?
- 自变量之间有什么关系?
多元方差分析需要满足前提假设:
- 各样本数据相互独立
- 每个分组内数据符合多元正态分布
- 每个分组内数据的协方差矩阵一致
但微生物群落丰度数据常常难以满足上述的条件,因此微生物数据的统计检验常用的是对数据分布没有要求的多元非参数方差检验:Anosim 和 Adonis。他们需要满足前提假设是:
- 每个对象的数据具有可交换性
- 可交换的对象(样品)彼此独立
- 每个样品的检测数据有一致的多变量分布(每组数据的离散程度相近)
零假设 :每组样本在其检测指标构成的检测空间中的中心点 (centroid)和离散度(dispersion)无差别,即组间存在差异。
Adonis 和 Anosim 都是对多维数据的非参数统计检验,都利用了置换检验(默认置换次数为 999)。不同点是应用的检验模型不同,Adonis 本质是基于 F 的统计量,常与 PCoA 配合使用,而 Anosim 的统计量是 R,常与 NMDS 配合使用。
当 Anosim 或 Adonis 计算的 p < 0.05,接受零假设,认为组间存在差异;否则拒绝零假设,认为组间不存在差异。
简而言之
Adonis 和 Anosim 都需要先计算一个统计量 F(或 R),然后通过样本置换获得一批置换样本,再计算一次统计量,通过一定 N 次的置换检验获得 N 个统计量,其中置换检验的统计量大于 F(或 R)的次数就是 p 值。
Anosim 和 Adonis 的具体说明请看下方的介绍。置换检验的说明请看下方 FAQ。
Anosim
Anosim [8](Analysis of similarities)是一种非参数检验方法,在生态学领域中被广泛使用。它首先计算相似性,然后将样品按相似性排秩,计算组间相似性均值 和组内相似性均值 ,然后按照下述公司计算 Anosim 的统计量 R:
:组间距离秩的均值
:组内距离秩的均值
:样本数
R 的取值范围值为 [-1, 1],R 趋近于 1 表示组内相似性高于组间相似性,R 趋近于 0 表示组间相似性和组内相似性无差异,R 小于 0 表示组间相似性大于组内相似性,R 越趋近 -1,样品分组错误的概率越大,您需要检查参与计算的样本是否被正确分组。
对统计量的假设检验利用了置换检验的结果,每次置换检验会得到一个 R',对于 N 次的置换检验,R > R' 的比例就是统计检验的 p 值。
Anosim 分析使用 R 语言 vegan 包中的 anosim 函数,常用的相似性矩阵是 Bray-Curtis 相似性矩阵。
Adonis
Adonis 又名 PERMANOVA [9](Permutational multivariate analysis of variance,置换多元方差分析)。PERMANOVA 采用数据置换的方式计算伪 F 值(pseudo F-ratio),它类似多元方差分析中的 F 统计量,计算公式为:
:伪 F 值(pseudo F-ratio)
:组内方差和
:组间方差和
:总样本数
:组内样本数
统计量 F 越大,表明组间差异越大。采用数据置换的方式计算 F 的显著性,比较随机置换数据获得的 F 是否高于或等于实际观测到的值。如果多于 5% 随机置换计算的 F 高于实际观测值,则表示不同组的样品之间不存在显著差异(p > 0.05)。
常见问题
Q:Bray-Curtis、JSD、欧氏距离有什么不同?如何选择?
A:欧式距离是对称指数,将双零数据视为相同的存在,另外两个是非对称指数,忽略双零数据。欧式距离的有一个特点是对数据的丰度数据的敏感性大于物种是否存在,并且欧式距离的计算方法决定了欧式距离的取值范围是无限大的。所以常用对于双零数据很多的物种丰度数据,选择 Bray-Curtis 和 JSD 会多一些,且 Bray-Curtis 相异性指数是最常用的指数。
Q:PCA、PCoA、PLS-DA、NMDS 的置信区间是什么?
A:如果有一个总体符合下图所示的正态分布,那么对于任意符合该分布的数据,其出现在区间 [-1.96, 1.96] 之间的概率为 95%。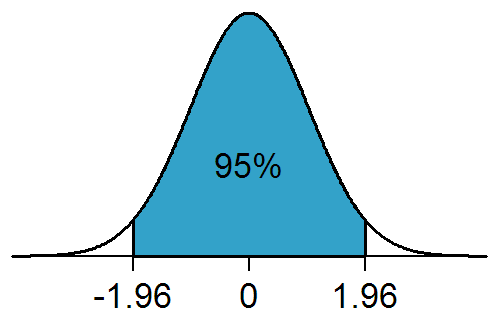
类似的,在 PCA、PCoA、PLS-DA 和 NMDS 中的置信椭圆标记了置信区间(Confidence Interval),是对样本分布的描述,这个区间表示的是对于任意给定的符合总体分布的样品,落在给定区间的概率是 95%。但是需要注意的是,我们通过采样获得的了数据总体的一部分,并用这个部分来表示数据的整体分布,因此样本数量会影响对总体分布分估计,并且样本数量越多对总体的描述越好。
Q:什么是置换检验?
A:通过进行实验和采样,获得了研究对象的部分数据,然后用这个数据来描述我们需要研究对象的整体。我们知道采样的样本量越大,对整体的描述就越准确,但实际实验经过程中,因为各种原因常得到的样本数量有限。当样本量很小,分布未知,如何用来描述总体呢,这个时候就需要用到置换检验。
置换检验(Permutation test)本质上是重抽样方法,它将全部(或部分)样本数据随机抽样排列,然后将抽样获得的样本统计量和实际观测到的样本统计量进行比较,通过大量的置换(R 中默认 999 次),计算置换后的统计量大于实际观测到的统计量的概率,即为置换检验 p 值,根据 p 值进行统计推断。这个方法最初由 Fisher 在 20 世纪 30 年代提出。
提示
因为置换检验进行的是随机抽样,所以多次置换检验的结果并不完全一致。
参考文献
Dodge, Y., Cox, D., & Commenges, D. (2006). The Oxford Dictionary of Statistical Terms. Oxford University Press. ↩︎
Whittaker, R. H. (1960). Vegetation of the Siskiyou Mountains, Oregon and California. Ecological Monographs, 30(3), 279–338. https://doi.org/10.2307/1943563 ↩︎
Legendre, P., & Legendre, L. (2012). Numerical ecology (Third English edition). Elsevier. ↩︎
Bray, J. R., & Curtis, J. T. (1957). An Ordination of the Upland Forest Communities of Southern Wisconsin. Ecological Monographs, 27(4), 325–349. https://doi.org/10.2307/1942268 ↩︎
Jolliffe, I. T., & Cadima, J. (2016). Principal component analysis: A review and recent developments. Philosophical Transactions of the Royal Society A: Mathematical, Physical and Engineering Sciences, 374(2065), 20150202. https://doi.org/10.1098/rsta.2015.0202 ↩︎
Hefner, R. (1959). Warren S. Torgerson, Theory and Methods of Scaling. New York: John Wiley and Sons, Inc., 1958. Pp. 460. Behavioral Science, 4(3), 245–247. https://doi.org/10.1002/bs.3830040308 ↩︎
Kenkel, N. C., & Orloci, L. (1986). Applying Metric and Nonmetric Multidimensional Scaling to Ecological Studies: Some New Results. Ecology, 67(4), 919–928. https://doi.org/10.2307/1939814 ↩︎
Clarke, K. R. (1993). Non-parametric Multivariate Analyses of Changes in Community Structure. Australian Journal of Ecology, 18(1), 117–143. https://doi.org/10.1111/j.1442-9993.1993.tb00438.x ↩︎
Stat, M., Pochon, X., Franklin, E. C., Bruno, J. F., Casey, K. S., Selig, E. R., & Gates, R. D. (2013). The Distribution of the Thermally Tolerant Symbiont Lineage (symbiodinium Clade D) in Corals from Hawaii: Correlations with Host and the History of Ocean Thermal Stress. Ecology and Evolution, 3(5), 1317–1329. https://doi.org/10.1002/ece3.556 ↩︎
